Une autre option pour un peu de programmation est le Indigo Toolkit, qui contient des bibliothèques Python, Java, .NET et C.
L'exemple Python suivant prend l'isomérique SMILES pour Albuterol, que j'ai obtenu de PubChem, et rend des structures 2D pour trois formes (une avec une stéréochimie non spécifiée et les deux stéréoisomères). Les structures individuelles sont étiquetées avec leurs CID PubChem respectifs.
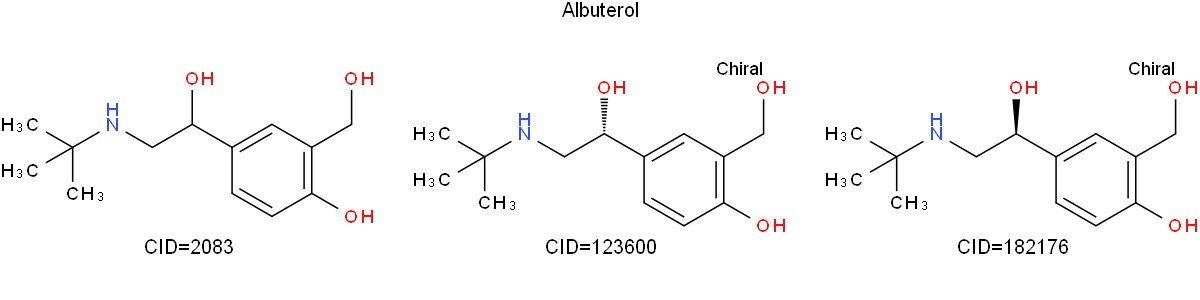
depuis import indigo * depuis indigo.renderer import * indigo = Indigo () renderer = IndigoRenderer (indigo) mols = {'2083': 'CC (C) (C) NCC (C1 = CC (= C (C = C1) O) CO) O', '123600': 'CC (C) (C) NC [C @@ H] (C1 = CC (= C (C = C1) O) CO) O ',' 182176 ':' CC (C) (C) NC [C @ H] (C1 = CC (= C (C = C1 ) O) CO) O '} array = indigo.createArray () for key in mols.keys (): print (key, mols [key]) mol = indigo.loadMolecule (mols [key]) s = "CID =" + clé mol.setProperty ("grid-comment", s) array.arrayAdd (mol) indigo.setOption ("render-comment", "Albuterol") indigo.setOption ("render-comment-position", "top") indigo.setOption ("render-grid-margins", "40, 10") indigo.setOption ("render-grid-title-offset", "5") indigo.setOption ("render-grid-title-property", "grid-comment") indigo.setOption ("rendu-fond-couleur", 1.0, 1.0, 1.0) indigo.setOption ("rendu-atome-couleur-propriété", "couleur") indigo.setOption ("rendu-coloration ", True) indigo.setOption (" render-image-size ", 1200, 300) renderer.renderGridToFile (array, None, 3, "grid.png")